 こんにちは、AnyTechの木村と申します。
こんにちは、AnyTechの木村と申します。
AnyTechにて、機械学習エンジニアやAIエンジニアといった役割にて、R&Dに日々従事しております。
この記事は、近年流行しているImageNetモデルを応用した異常検知手法について、リサーチをする機会がありましたので、小職の備忘も兼ねまして、その解説をさせて頂くものです。
目次
シリーズ
- 第1回:導入と転移学習
- 第2回:DN2(Deep Nearest Neighbor Anomaly Detection)
- 第3回:SPADE(Sub-Image Anomaly Detection with Deep Pyramid Correspondences) ◀ 今回ここ
- 第4回:PaDiM(a Patch Distribution Modeling Framework for Anomaly Detection and Localization)
- 第5回:PatchCore(Towards Total Recall in Industrial Anomaly Detection)
はじめに
第2回に引き続き、近年流行しているImageNetモデルを応用した異常検知手法について、解説をさせて頂きます。
今回は、SPADE(Sub-Image Anomaly Detection with Deep Pyramid Correspondences)についての解説となります。
SPADEは、DN2で突かれた間隙から、その隙間を上手くこじ開けたアルゴリズムだと思います。
以下の画像のようなイメージでしょうか。笑
また、ベースのモデルとして機能するImageNetモデルについて、それをwhitebox的に扱うことで発展を実現しています。
その意味では、SPADEを抑えることで、CNNに対する原理理解も深まるかと思います。
今回の記事で、上手くその辺りを伝えたいと思います。

先ずはザックリ、アルゴリズム概要
SPADEは、前段として、DN2を実施することで、画像レベルでの異常検知を行います。
そして、後段として、pixelレベルの異常位置セグメンテーションを行います。
この後段が、SPADEの新規性になります。
pixelレベルの異常位置セグメンテーションのために、クエリ画像と、DN2でのKNN実施によって獲得したK個のご近所さん画像より、縦横の概念を持った中間層の特徴テンソルの抽出を行います。
DN2では、最終層の特徴ベクトルしか用いませんでしたが、SPADEでは、中間層の特徴テンソルも用います。
中間層の階層別に、クエリ画像の特徴上の各pixelについて、K個のご近所さん画像から得られる特徴上の全pixelとのKNNによる距離計算を行います。
そうして得られた距離を、クエリ画像の特徴上の各pixelにおける異常スコアとすることで、異常スコアマップを構築します。
後は、異常スコアマップを元の解像度に拡大し、階層別に求めたそれらの平均を取った上で、ガウシアンフィルタで滑らかにして、1枚の異常スコアマップを仕上げます。
ここで、各異常スコアは相対的なものとなりますので、評価データにて最適な閾値を探索し、それより大きいか小さいかで、pixelレベルで異常か正常かを判断する形となります。
先ず、DN2で画像レベルの異常検知を実施して…

次に、SPADEのpixelレベルの異常位置セグメンテーションを実施する

次にじっくり、アルゴリズム詳細
ここからはじっくりと、SPADEの詳細を紹介させて頂こうと思います。
SPADEの論文において、DN2での課題点についての記載があります。
それは、「DN2は、入力クエリ画像の正常/異常判定はできるのだけども、画像上の異常位置や、モデルは画像上のどこを見て異常と判断しているのかが表現できない」というものです。
SPADEは、DN2を発展させて、その点を改善させたものとなります。
つまり、画像レベルでの異常検知を行うDN2に加えて、pixelレベルでの異常位置セグメンテーションの機能を加えたものが、SPADEとなっています。
尚、異常位置セグメンテーションについては、例えば、いわゆるPost-hocのExplainable AIによって、DN2の要因分析を行うことで実現も可能でしょう。
ここで、Post-hocとは、後付けという意味です。(同僚に教えてもらった便利な表現🙏)
例えば、Post-hocのExplainable AIの1つに、LIMEという要因分析手法がありますが、それであれば異常位置セグメンテーションの実施が可能です。
尚、LIMEの詳細については、以下の記事が分かりやすいかと思いますので、参考までに。
Explainable AI:LIMEを用いた判断根拠の可視化 - kentaPtの日記
以下のスライドも非常に分かりやすいです。

しかし、LIMEのような後付の要因分析を行おうとすると、判断根拠を調べる為に複数回のモデル計算を要する等、処理がかなり重たくなってしまいます。
具体的には、クエリ画像の一部分を色々なバリエーションで隠しながら、その予測挙動を伺うことで、要因分析を行う形となります。
そうして、判断にクリティカルな箇所を探す為です。
実用などを考えた場合には、この計算負荷は避けたいところです。
SPADEでは、そんな異常位置セグメンテーションについて、比較的ストレートフォワードでリーズナブルなアプローチを提案してくれています。
尚、SPADEというアルゴリズムは、「①画像単位の異常スコア算出」と「②画像上の異常位置セグメンテーション」という、2段構えの構成となっており、それらはアルゴリズムが概ね独立しています。
2者の間には若干の関連があるのみで、ほぼ別アルゴリズムと言っても過言ではありません。
そして、なんと、「①画像単位の異常スコア算出」に関しては、DN2を実施しています。
新たな工夫等は全くなく、DN2そのものを適用しています。
つまり、「SPADE = DN2 + ②画像上の異常位置セグメンテーション」という内訳となっています。
その為、ここまで読んで下さった方は、既にSPADEというアルゴリズムの半分を理解して頂いている次第です。
また、データベース別にSOTAをまとめてくれているサイト「Paper With Code」に、MVTecのSOTAランキングも存在するのですが、そこにSPADEもランクインをしています。
そして、記載の精度には、「Detection AUROC」と「Segmentation AUROC」という2つの指標が存在しており、これらが「Detection AUROC=①画像単位の異常スコアの精度」と「Segmentation AUROC=②画像上の異常位置セグメンテーションの精度」という意味になります。
その為、SPADEの「Detection AUROC」は、DN2のそれでもあります。
尚、SPADEにおける「①画像単位の異常検知」と「②画像上の異常位置セグメンテーション」は、概ねそれぞれが独立したアルゴリズムになっていますが、後続のPaDiMとPatchCoreではそれらは密に関連しています。
詳細は後述しますが、大まかには、クエリ画像に対して「②」を先に実施し、そこで得られたpixelレベルでの異常スコアの内の最大値を、「①」における画像レベルでの異常スコアとする形です。
SPADEにおいても、場合によっては、「②→①」という流れで、画像レベルの異常検知を実施しても良いかもしれません。
さて、ここからは具体的に、SPADEのアルゴリズムについて、主に「②画像上の異常位置セグメンテーション」を中心に、解説をさせて頂こうと思います。
以下は、論文に記載されている説明を、概ねシンプルに和訳しつつ、補足を加えたものとなります。
SPADEのアルゴリズムは、以下3段構成となっている。
- 画像の特徴抽出
- K個のご近所さんインデックスの取得
- 中間層特徴のピラミッド対応による異常位置セグメンテーション
第1段階として、画像の縦横概念を持った中間層の特徴テンソル、及び、グローバルプーリング後等の最終層の特徴ベクトルを取得する。
中間層の特徴テンソルは、pixelレベルの異常位置セグメンテーションに用いる。
最終層の特徴ベクトルは、DN2の実施、及び、K個のご近所さんインデックスの取得に用いる。
尚、ImageNetモデルのResNet(※特にWideResNetと別途後述)を、特徴抽出器として使用する。
データが多ければ、「self-supervised feature learning」というオプションも魅力的ですが、Bergmanらの分析によれば、それはクラス識別目的向きであり、異常検知目的ではImageNetモデルの方が性能が優るとのこと。
特徴抽出器を とし、与えられた画像
に対して、抽出された特徴量を
とする。

尚、論文上は、最終層の特徴ベクトルを 、中間層の特徴テンソルを
と表現する。
は、任意のpixel、即ち、特徴抽出元の位置を意味するもの。
の引数にそれが含まれていたら、中間層の特徴テンソルを意味すると捉える。
この特徴抽出を、学習データ(全て正常データ)について実施する。
この算出までが第1段階で、いわば学習に当たる部分となる。
尚、運用時には、学習データから抽出した特徴を保存しておいたものをロードします。
その為、運用時には、クエリ画像のみから特徴抽出を行う。
第2段階は、KNNを実施して、DN2の実施と、K個のご近所さんのインデックス取得とを両立する。
DN2では、KNNによって算出された距離だけを用いましたが、SPADEでは、そのご近所さんのインデックス、即ち、そのご近所さんとの距離以外の情報も用いる。
この段階にて、KNNの実施に用いるのは、最終層の特徴ベクトル。
クエリ画像 から抽出される特徴
として、特徴ギャラリーから得られるK個のご近所さんを
とする。
そのK個のご近所さんの、最終層の特徴ベクトルを
と表現する。
つまり、ここでの は、最終層の特徴ベクトルがK本含まれているイメージとなる。
そうして得たK本の最終層の特徴ベクトルと、クエリ画像から得た1本の最終層の特徴ベクトルとの距離を、K個算出し、その平均を求める。
これは、DN2の実施となる。

第3段階は、画像の位置合わせによるサブ画像異常検出を実施する。
「画像の位置合わせによるサブ画像異常検出=Sub-image Anomaly Detection via Image Alignment(SPADE)」であり、ここがアルゴリズムの本懐となる。
ここで達成したいことは、以下。
- 画像レベルで異常と判定されたものについて、1つ、または、複数の異常pixel位置を特定し、原因を切り出す
- 画像レベルで誤って異常と分類された場合について、異常と分類された画素が無いことを示す
また、一般的に、テスト画像と正常画像との位置合わせを考えた場合、それらの差分から異常画素を検出することを考えると、その素朴な方法には幾つかの欠点がある。
- テスト画像が複数の正常な部分から構成されている場合(つまり、ほぼ正常ということが言いたそう)、比較対象には特に正常度合いの高い画像を用意する必要があるが、その用意に失敗してしまう可能性がある
- データセットが小さかったり(多様性の網羅性が低い)、複雑な変化をするオブジェクト(弾性体/非剛体であったりで、pixel位置依存性が低かったり)の場合、テスト画像とあらゆる点で類似している正常な画像を見つけることができず、誤検出を引き起こす可能性がある
- 画像の差分を計算する際、使用する損失関数に非常に敏感である(SADとSSDで結果が結構変わる等か)
これらの欠点を克服するために、複数画像の対応付けを行う手法を、私達は提案する。
特徴抽出器 を用いて、画素位置
毎に、deepな特徴を抽出することで、それを実現する。
実現の手順としては、先ず、K個のご近所さん画像の、全ての画素位置における特徴を集め、特徴ギャラリー を構築する。
次に、クエリ画像から、各画素位置毎の特徴 を得る。
最後に、クエリ画像の各画素位置毎の特徴と、K個のご近所さん画像の全画素位置における特徴ギャラリーとでKNNを実施し、そのKNN距離をクエリ画像の各画素位置 毎に異常スコアとする。
式表現にすると以下。

そして、与えられた閾値 に対して、
となる場合、つまり、K個のご近所さんから構築した特徴ギャラリー
(これらは皆、正常画像の各画素
からなる)の中から、クエリ画像の各画素位置毎の特徴
と近似度の高い
(
は任意という意味)が見つからない場合、その画素が異常と判定される。
ここで、特徴抽出器ついて掘り下げる。
前提として、画像の正常部分と異常部分とを分別するには、密な対応関係によるアライメントが有効となる。
そして、アライメントを効果的に行うためには、マッチングのための特徴量を決定する必要がある。
本手法では、その特徴に、deep ResNet CNNの特徴を用いている。
そのResNet特徴ですが、それはピラミッド形状となっている。
画像のピラミッドと同様に、初期の層(浅めの層)は、より少ないコンテキストがエンコードされた、高解像度の特徴となる。
終盤の層(深めの層)は、より多くのコンテキスト(チャンネルが多い、デプスが厚い)がエンコードされた、低解像度の特徴となる。
効果的なアライメントを行うために、それらを特徴ピラミッドの層の深さ毎、及び、画像上のpixel位置毎に特徴記述する。
尚、この特徴には、細かい局所的なものと、荒い大局的なものとが、両方含まれている。
これにより、技術的に困難で脆弱である、画像全体の位置合わせを行うのではなく、画像上のpixel位置毎に、クエリ画像の特徴と、K個のご近所さん画像(全て正常画像)の特徴との、KNN対応付けが可能となる。
この方法はpixel依存がない為、スケーラブルで実用向けとなる。
最後の補足として、実装の詳細を示す。
- 実験においては、ImageNetで事前に学習したWide-ResNet50×2の特徴抽出器を使用
- MVTecの画像は256×256にリサイズした後、224×224にトリミング
- 必要に応じてcv2.INTERAREAを使用してリサイズを実施(※一般に、その方がキレイに縮小されるとのこと:ref)
- 特に指定しない限り、ResNetからの特徴は、第1ブロック(56 × 56)、第2ブロック(28 × 28)、第3ブロック(14 × 14)の最後に、すべて等しい重みで使用(※層別にインパクトの強弱といった重み付けはしていない、という意味と思われる)
- 実験におけるKNN実施については、MVTecでのご近所さん画像取得についてはK=50とし、pixel位置毎の対応検索については、k=1とした
- pixel単位の異常スコアを求めた後、最終的にはそれを
のガウシアンフィルタで平滑化した
以上が、論文に沿ったアルゴリズムの説明となります。
DN2をpixel単位にも展開させるような発想で、異常位置のセグメンテーションも実現してしまっているという、秀逸な発展論文になっているかと思います。
尚、補足になりますが、最終層の特徴ベクトルは、画像全体の特徴をベクトル化したものである為、画像上のどの位置にどういう特徴があるか、という情報が直接的には残っていません。(間接的には、LIME等で特定できる可能性がある。)
その為、「②画像上の異常位置セグメンテーション」をストレートフォワードに行うためには、例えば、中間層の特徴が必要となります。
中間層の特徴は、いわゆるテンソルという形状をしています。
1次元の配列に数値が並んでいるベクトルに対して、テンソルは3次元以上の配列に数値が並んでいるものとなります。
実際に、中間層特徴テンソルのプログラム上の管理としては、 (Batch数, 特徴チャンネル数, 縦pixel数, 横pixel数) という形で管理され、4次元のテンソルとなっています。
或いは、3次元のテンソルが、Batch数個だけ管理されている形です。
モデルを通過する過程で、或いは、モデル計算の途中で、入力の画像 (Batch数, RGBチャンネル数, 縦pixel数, 横pixel数) が変化したものが、 (Batch数, 特徴チャンネル数, 縦pixel数, 横pixel数) となる形です。
一般に、中間特徴の 縦pixel数 横pixel数 は、入力画像のそれよりも小さくなっている傾向があります。
また、中間特徴の 特徴チャンネル数 は、入力画像のそれ(概ねRGBの3チャンネル)よりも大きくなっている傾向があります。
最終的なベクトルは、いわば 縦pixel数 = 1 横pixel数 = 1 という状態であり、つまりは、モデル計算が深く進んでいく程に、段々と画像の縦横の概念、即ち、空間情報が圧縮されていく次第となっています。
イメージとしては以下となります。

尚、補足として、以下のような縦横pixelが小さくならないモデルも存在します。

以下のように、行ったり来たりのモデルも存在します。
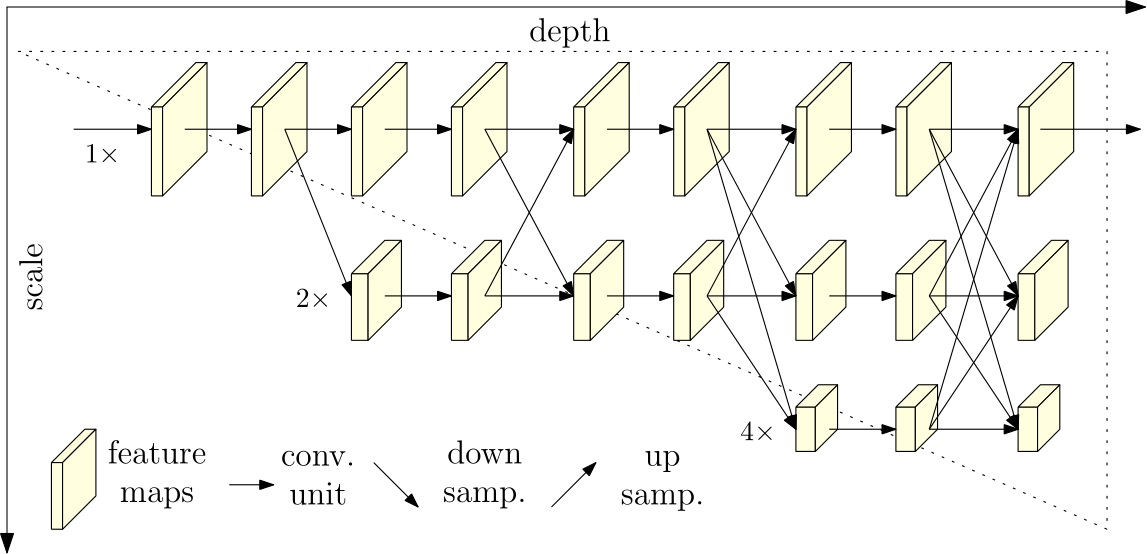
面白いですね。w
この辺りは、受容野などを考慮に入れつつ、適切と思われるモデルを選択し、実験してみる必要があります。
さて、それでは、より具体的にイメージを掴むために、実装をしていきましょう。
ここでは、SPADEのGithubリポジトリの1つであるSPADE-pytorchに倣って、特徴抽出器として用いるモデルに torchvision.models.wide_resnet50_2 を用います。
その仕掛け作りのコードは以下となります。
import torch
import torchvision.models as models
device = torch.device('cuda:0') # torch.device('cpu')
model = models.wide_resnet50_2(weights=models.Wide_ResNet50_2_Weights.IMAGENET1K_V1)
model.eval()
model.to(device)
print('model =', model)
# set model's intermediate outputs
outputs = []
def hook(module, input, output):
outputs.append(output.clone().detach().cpu().numpy())
model.layer1[-1].register_forward_hook(hook)
model.layer2[-1].register_forward_hook(hook)
model.layer3[-1].register_forward_hook(hook)
model.avgpool.register_forward_hook(hook)
DN2の実装時よりも、hookする層が増えています。
増えたhook先が、先述のアルゴリズム説明にある中間層特徴となります。
model.layer1[-1] は、結構浅めの層で、入力が例えば、 (Batch数:1, RGBチャンネル数:3, 縦pixel数:224, 横pixel数:224) だった場合に、 (Batch数:1, 特徴チャンネル数:256, 縦pixel数:56, 横pixel数:56) という形状の特徴を出力する層となります。
model.layer2[-1] は、正に中間層という感じで、先程と同じ入力に対して、 (Batch数:1, 特徴チャンネル数:512, 縦pixel数:28, 横pixel数:28) という形状の特徴を出力します。
model.layer3[-1] は、深めの層で、同入力に対し、 (Batch数:1, 特徴チャンネル数:1024, 縦pixel数:14, 横pixel数:14) という形状の特徴を出力します。
よって、上記のhookによって、画像上のpixel単位の異常位置セグメンテーションを行うための特徴抽出について、準備ができた次第です。
ちなみに、ロードするモデルの重みですが、DN2の際に採用していた weights=models.Wide_ResNet50_2_Weights.DEFAULT) 、即ち、 weights=models.Wide_ResNet50_2_Weights.IMAGENET1K_V2) よりも、どうも weights=models.Wide_ResNet50_2_Weights.IMAGENET1K_V1) の方が精度が高いようで、こちらを採用しています。
論文との精度比較にて、SPADEの著者も、こちらの重みを用いているものと思います。
SPADE論文に記載されているfeedforwardのための前処理方法が、torchvisionに書かれている前処理とも合致しています。
wide_resnet50_2 — Torchvision main documentation
次に、DN2の時と同じ様に、Cats vs Dogsを読み込みます。
以下コードでファイル名を取得します。
import os
path = './dogs-vs-cats/train/'
files = os.listdir(path)
files_cat = [os.path.join(path, f) for f in files
if (os.path.isfile(os.path.join(path, f)) &
('cat.' in f) & ('.jpg' in f))]
files_dog = [os.path.join(path, f) for f in files
if (os.path.isfile(os.path.join(path, f)) &
('dog.' in f) & ('.jpg' in f))]
files_cat = sorted(files_cat)
files_dog = sorted(files_dog)
print('len(files_cat) =', len(files_cat))
print('files_cat[:10] =\n', files_cat[:10])
print()
print('len(files_dog) =', len(files_dog))
print('files_dog[:10] =\n', files_dog[:10])
次に、取得したファイル名を、学習データと評価データに分けます。
import random
import numpy as np
N_cat_train = 3000
N_cat_val = 1000
N_dog = 1000
random.seed(0)
files_cat = random.sample(files_cat, (N_cat_train + N_cat_val))
files_dog = random.sample(files_dog, N_dog)
files_train = np.array(files_cat[:N_cat_train])
files_val = np.array(files_cat[N_cat_train:] + files_dog)
y_val = np.concatenate([np.zeros([N_cat_val]),
np.ones([N_dog])], axis=0).astype(np.int16)
print('len(files_train) =', len(files_train))
print('len(files_val) =', len(files_val))
import matplotlib.pyplot as plt
import japanize_matplotlib
plt.figure(figsize=(10, 4), dpi=100)
plt.plot(y_val, linewidth=5, alpha=0.5)
plt.grid()
plt.show()
尚、学習データと評価データの件数ですが、DN2の時よりも減らす必要があるかもしれません。
中間層の特徴も保持することとなり、RAMが逼迫する為です。
このSPADEのRAM逼迫感は、後続の論文でも指摘がされる要改善点ともなっています。
異常位置のセグメンテーションをする為の代償とも言える課題感です。
という訳で、SPADEについては、学習データの猫画像の枚数を3,000枚としました。
評価データの件数は変わらず犬猫各1,000枚です。
ひょっとすると、コードを追いかけて下さっている方の環境においては、この課題感がもっとシビアに現れるかもしれません。
その際は、学習データ、及び、評価データの件数を見直して頂けたらと思います。
さて、学習データのファイル名と、評価データのファイル名が取得できたら、次には特徴の抽出を実施します。
繰り返しになりますが、SPADEを実施する際には、中間層の特徴テンソルを取得します。
また、DN2の実施と、異常位置セグメンテーション用の特徴ギャラリー生成元となるご近所さん探索のために、最終層の特徴ベクトルも取得します。
また、DN2の実装では特徴抽出の際に、1画像ずつをfeedforwardにかけていましたが、当該SPADE実装では処理速度を上げるために、複数画像ずつfeedforwardにかけるようにしてみましょう。
以下が、学習データより特徴を抽出するコードとなります。
import cv2
from tqdm.notebook import tqdm
N_batch = 200
outputs = []
x_batch = []
img_train = []
img_prep_train = []
for file in tqdm(files_train):
img = cv2.imread(file)[..., ::-1] # BGR2RGB
img_prep = cv2.resize(img, (224, 224))
x = img_prep
x = x / 255
x = x - np.array([[[0.485, 0.456, 0.406]]])
x = x / np.array([[[0.229, 0.224, 0.225]]])
x = torch.from_numpy(x.astype(np.float32)).unsqueeze(0).permute(0, 3, 1, 2)
x = x.to(device)
x_batch.append(x)
if (len(x_batch) == N_batch) | (file == files_train[-1]):
with torch.no_grad():
_ = model(torch.vstack(x_batch))
x_batch = []
img_train.append(img)
img_prep_train.append(img_prep)
img_prep_train = np.stack(img_prep_train)
f1_train = np.vstack(outputs[0::4])
f2_train = np.vstack(outputs[1::4])
f3_train = np.vstack(outputs[2::4])
fl_train = np.vstack(outputs[3::4]).squeeze(-1).squeeze(-1)
print('len(img_train) =', len(img_train))
print('img_prep_train.shape =', img_prep_train.shape)
print('f1_train.shape =', f1_train.shape)
print('f2_train.shape =', f2_train.shape)
print('f3_train.shape =', f3_train.shape)
print('fl_train.shape =', fl_train.shape)
次に、評価データより特徴を抽出するコードです。
outputs = []
x_batch = []
img_val = []
img_prep_val = []
for file in tqdm(files_val):
img = cv2.imread(file)[..., ::-1] # BGR2RGB
img_prep = cv2.resize(img, (224, 224))
x = img_prep
x = x / 255
x = x - np.array([[[0.485, 0.456, 0.406]]])
x = x / np.array([[[0.229, 0.224, 0.225]]])
x = torch.from_numpy(x.astype(np.float32)).unsqueeze(0).permute(0, 3, 1, 2)
x = x.to(device)
x_batch.append(x)
if (len(x_batch) == N_batch) | (file == files_val[-1]):
with torch.no_grad():
_ = model(torch.vstack(x_batch))
x_batch = []
img_val.append(img)
img_prep_val.append(img_prep)
img_prep_val = np.stack(img_prep_val)
f1_val = np.vstack(outputs[0::4])
f2_val = np.vstack(outputs[1::4])
f3_val = np.vstack(outputs[2::4])
fl_val = np.vstack(outputs[3::4]).squeeze(-1).squeeze(-1)
print('len(img_val) =', len(img_val))
print('img_prep_val.shape =', img_prep_val.shape)
print('f1_val.shape =', f1_val.shape)
print('f2_val.shape =', f2_val.shape)
print('f3_val.shape =', f3_val.shape)
print('fl_val.shape =', fl_val.shape)
これで、処理に必要な特徴が抽出されました。
次には、DN2の実施と、評価データとのご近所さん取得とに向けて、最終層の特徴ベクトルによるKNNを実施します。
学習データにおける最終層の特徴ベクトルを特徴ギャラリーに登録して、各評価データの最終層の特徴ベクトルでもって、評価データに近い学習データのご近所さんをK個探します。
以下が、それを実践するコードとなります。
import faiss
d = fl_train.shape[1]
index = faiss.GpuIndexFlatL2(faiss.StandardGpuResources(),
d,
faiss.GpuIndexFlatConfig())
index.add(fl_train)
print('index =', index)
print('index.ntotal =', index.ntotal)
k = 5 # we want to see 5 nearest neighbors
D_val, I_val = index.search(fl_val, k)
print('D_val.shape =', D_val.shape)
print('I_val.shape =', I_val.shape)
plt.figure(figsize=(10, 6), dpi=100, facecolor='white')
plt.subplot(1, 2, 1)
plt.imshow(D_val, aspect='auto')
plt.colorbar()
plt.title('KNN distanse')
plt.subplot(1, 2, 2)
plt.imshow(I_val, aspect='auto')
plt.colorbar()
plt.title('KNN index')
plt.show()
plt.figure(figsize=(10, 8), dpi=100, facecolor='white')
plt.subplot(2, 1, 1)
plt.scatter(np.where(y_val == 0)[0],
np.mean(D_val[y_val == 0], axis=1), alpha=0.5, label='猫画像')
plt.scatter(np.where(y_val == 1)[0],
np.mean(D_val[y_val == 1], axis=1), alpha=0.5, label='犬画像')
plt.grid()
plt.legend()
plt.subplot(2, 1, 2)
plt.hist(np.mean(D_val[y_val == 0], axis=1), alpha=0.5, bins=50, label='猫画像')
plt.hist(np.mean(D_val[y_val == 1], axis=1), alpha=0.5,
bins=int(50*(N_dog/N_cat_val)), label='犬画像')
plt.grid()
plt.legend()
plt.show()
acc_list = []
thresh_list = np.arange(0, np.max(np.mean(D_val, axis=1))+1e-10,
np.max(np.mean(D_val, axis=1))/50)
for thresh in thresh_list:
acc = np.mean(np.concatenate([(np.mean(D_val[y_val == 0], axis=1) < thresh),
(np.mean(D_val[y_val == 1], axis=1) > thresh)]))
acc_list.append(acc)
acc_list = np.array(acc_list)
plt.figure(figsize=(10, 4), dpi=100, facecolor='white')
plt.plot(thresh_list, acc_list, alpha=0.5, linewidth=3)
plt.scatter(thresh_list, acc_list, alpha=0.5, s=30)
plt.grid()
plt.title('np.max(acc_list) = %.3f, thresh_list[np.argmax(acc_list) = %.2f' %
(np.max(acc_list), thresh_list[np.argmax(acc_list)]))
plt.show()



先ず、DN2は上手くいきました。
これにて、SPADEというアルゴリズムの2段構成の前段「①画像単位の異常スコア算出」が完了した形になります。
残る、後段の「②画像上の異常位置セグメンテーション」をここから実施していきます。
「②画像上の異常位置セグメンテーション」の実施に当たっては、上記コードにおける変数 I_val が重要となります。
ちなみに、この変数 I_val はご近所さんの存在そのものを示すインデックスであり、SPADEの前段「①画像単位の異常スコア算出」、つまり、DN2の実施に際しては、不要なものとなります。
上記コードでも、D_val, I_val = index.search(fl_val, k) というコードにて左辺で受け取った後、それを右辺で使用することをせずに、DN2のアルゴリズムが完結しています。
DN2は距離の値があれば、それで実現ができる為です。
尚、改めてですが、 D_val には各評価データ毎に、学習データのご近所さんK個との距離が格納されています。
その為、 D_val 変数の形状は、 (評価データ件数, K) となっています。
I_val 変数の形状の同じ、 (評価データ件数, K) となっていて、内容としては、各評価データ毎の、学習データのご近所さんK個のインデックスが格納されています。
尚、 D_val も I_val も、近い順に並んでいます。
具体的には、例えば、 I_val[10, 0] には、10番インデックスの評価データと最も近い学習データのインデックスが格納されています。
そして、それらの距離は D_val[10, 0] になるという寸法です。
同様に、10番インデックスの評価データと、2番目に近い学習データのインデックスは I_val[10, 1] に格納されていて、それらの距離は、 D_val[10, 1] となります。
さて、 I_val 変数を用いて、SPADEの後段「②画像上の異常位置セグメンテーション」を実施します。
その初手としまして、先述のアルゴリズム説明に沿って、各評価データについて、中間層の深さ別に、pixel毎の特徴ベクトルによる特徴ギャラリーを収集します。
尚、分かりやすさから、ここでの説明では、特定の評価データに注目をして、それを行っていこうと思います。
先ず、特に異常と判断された猫画像について確認をし、めぼしい対象をピックアップします。
そして、異常と判断される画像位置が、我々人間の一般的な認識に合うかどうかを確認しようと思います。
猫画像の中で、特に異常と判断されたもの、即ち、ご近所さんK個との距離の平均が大きかったものを見てみます。
以下のコードで、それを確認します。
idx_miss_cat = np.argsort(-np.abs(np.mean(D_val[y_val == 0], axis=1)))
for j_miss, i_miss in enumerate(idx_miss_cat[:10]):
img = [img_val[i] for i in np.where(y_val == 0)[0]][i_miss]
img_prep = img_prep_val[y_val == 0][i_miss]
plt.figure(figsize=(10, 6), dpi=100, facecolor='white')
plt.subplot(1, 2, 1)
plt.imshow(img)
plt.subplot(1, 2, 2)
plt.imshow(img_prep)
plt.title('rank:%d, D:%.3f' %
((j_miss + 1), np.mean(D_val[y_val == 0], axis=1)[i_miss]))
plt.show()
猫画像の内、算出距離の大きかった画像TOP1

猫画像の内、算出距離の大きかった画像TOP2

猫画像の内、算出距離の大きかった画像TOP3

猫画像の内、算出距離の大きかった画像TOP4

猫画像の内、算出距離の大きかった画像TOP5

猫画像の内、算出距離の大きかった画像TOP6

猫画像の内、算出距離の大きかった画像TOP7

猫画像の内、算出距離の大きかった画像TOP8

猫画像の内、算出距離の大きかった画像TOP9

猫画像の内、算出距離の大きかった画像TOP10

ここで、以下のクエリ画像に着目して、SPADEの「②画像上の異常位置セグメンテーション」がどのように行われるのかを見ていきます。

KNNのKは5個としていましたので、5個のご近所さんインデックスが I_val 変数より取得できます。
そのインデックスより、注目画像とのご近所さん画像を確認します。
尚、右側画像の上部に記載されている数値は、着目のクエリ画像との距離になります。
また、画像確認のついでに、中間層の特徴テンソルもピックアップします。
上記実践を行うのが、以下コードとなります。
i_miss = idx_miss_cat[9]
print('i_miss =', i_miss)
img = [img_val[i] for i in np.where(y_val == 0)[0]][i_miss]
img_prep = img_prep_val[y_val == 0][i_miss]
plt.figure(figsize=(10, 6), dpi=100, facecolor='white')
plt.subplot(1, 2, 1)
plt.imshow(img)
plt.subplot(1, 2, 2)
plt.imshow(img_prep)
plt.title('D:%.3f' % np.mean(D_val[y_val == 0][i_miss]))
plt.show()
for i_k, i_nn in enumerate(I_val[y_val == 0][i_miss]):
print('i_nn =', i_nn)
img = img_train[i_nn]
img_prep = img_prep_train[i_nn]
plt.figure(figsize=(10, 6), dpi=100, facecolor='white')
plt.subplot(1, 2, 1)
plt.imshow(img)
plt.subplot(1, 2, 2)
plt.imshow(img_prep)
plt.title('D:%.3f' % D_val[y_val == 0][i_miss][i_k])
plt.show()
f1_query = f1_val[y_val == 0][[i_miss]]
f2_query = f2_val[y_val == 0][[i_miss]]
f3_query = f3_val[y_val == 0][[i_miss]]
f1_neighbor = f1_train[I_val[y_val == 0][i_miss]]
f2_neighbor = f2_train[I_val[y_val == 0][i_miss]]
f3_neighbor = f3_train[I_val[y_val == 0][i_miss]]
print('f1_query.shape =', f1_query.shape)
print('f2_query.shape =', f2_query.shape)
print('f3_query.shape =', f3_query.shape)
print()
print('f1_neighbor.shape =', f1_neighbor.shape)
print('f2_neighbor.shape =', f2_neighbor.shape)
print('f3_neighbor.shape =', f3_neighbor.shape)
score_maps = []
学習データの猫画像の内、クエリ画像との距離の小さかった画像TOP1

学習データの猫画像の内、クエリ画像との距離の小さかった画像TOP2

学習データの猫画像の内、クエリ画像との距離の小さかった画像TOP3

学習データの猫画像の内、クエリ画像との距離の小さかった画像TOP4

学習データの猫画像の内、クエリ画像との距離の小さかった画像TOP5

次に、このK個のご近所さんの中間層特徴から、特徴ギャラリーを作成します。
クエリ画像のpixel毎の特徴 との総当り距離計算用に作成をする、ご近所さんK個の各pixel毎の特徴
のことです。
また、クエリ画像の各pixel毎の特徴 の整理も行います。
それでは、最初は浅めの層の特徴 (Batch数:1, 特徴チャンネル数:256, 縦pixel数:56, 横pixel数:56) から異常位置セグメンテーションを行いします。
以下コードが、その実践です。
尚、このコードですが、SPADE-pytorchリポジトリよりの引用であり、これは小職がPRを出している修正版のコードとなっています。
# https://github.com/mucunwuxian/SPADE-pytorch/blob/master/src/main.py#L130
# construct a gallery of features at all pixel locations of the K nearest neighbors
topk_feat_map = torch.from_numpy(f1_neighbor)
test_feat_map = torch.from_numpy(f1_query)
# adjust dimensions to measure distance in the channel dimension for all combinations
feat_gallery = topk_feat_map.transpose(1, 2).transpose(2, 3) # (K, C, H, W) -> (K, H, W, C)
feat_gallery = feat_gallery.flatten(0, 2) # (K, H, W, C) -> (KHW, C)
feat_gallery = feat_gallery.unsqueeze(1).unsqueeze(1) # (KHW, C) -> (KHW, 1, 1, C)
test_feat_map = test_feat_map.transpose(1, 2).transpose(2, 3) # (K, C, H, W) -> (K, H, W, C)
print('topk_feat_map.shape =', topk_feat_map.shape)
print('test_feat_map.shape =', test_feat_map.shape)
print('feat_gallery.shape =', feat_gallery.shape)
こうして特徴の整理ができましたら、総当たりの距離計算を実施します。
上記コードでの整理は、以下コードで使用される torch.pairwise_distance メソッドを用いるために行ったものとなっています。
また、この torch.pairwise_distance の計算に対して、多数の特徴が含まれる特徴ギャラリーから、100件ずつが小出しにて、計算対象とされています。
これは、あまりに多くデータを突っ込みすぎると、GPUのRAMがパンクするためにそうしているものとなります。
# calculate distance matrix
dist_matrix_list = []
for d_idx in tqdm(range(feat_gallery.shape[0] // 100 + 1)):
dist_matrix = torch.pairwise_distance(feat_gallery[d_idx*100:d_idx*100+100],
test_feat_map)
dist_matrix_list.append(dist_matrix)
dist_matrix = torch.cat(dist_matrix_list, 0)
print('dist_matrix.shape =', dist_matrix.shape)
この計算結果から、 (特徴ギャラリーにある各pixel毎の特徴ベクトル本数:15,680, クエリ画像からの特徴テンソルの縦サイズ:56, クエリ画像からの特徴テンソルの横サイズ:56) という結果が得られます。
この結果については、しっかりと整理をしましょう。
クエリ画像からの特徴テンソルには、縦サイズ:56pixel、横サイズ:56pixelに、それぞれ256次元の特徴ベクトルが保持されている形となっています。
対して、K=5個のご近所さんからの特徴ギャラリーには、 K:5 × 縦サイズ:56pixel × 横サイズ:56pixel = 15,680本 の特徴ベクトルが保持されている形となっています。
これらを総当りで距離計算するので、 (特徴ギャラリーにある各pixel毎の特徴ベクトル本数:15,680, クエリ画像からの特徴テンソルの縦サイズ:56, クエリ画像からの特徴テンソルの横サイズ:56) という結果が得られる形となります。
論文の説明から、ここでやっていることは、K個のご近所さんの局所的な画像パッチに対して、クエリ画像の局所的な画像パッチと類似する対象を探索しにいっている形となっています。
即ち、K個のご近所さんの局所的な画像パッチの中に、クエリ画像の局所的な画像パッチと似たものがあれば、距離計算結果が小さな値にて返されることとなります。
逆に、K個のご近所さんの局所的な画像パッチの中に、クエリ画像の局所的な画像パッチと似たものがなければ、そのパッチによって計算された距離は、その全てが大きな値となってしまいます。
つまり、その全てが大きな値となってしまう画像パッチが、異常位置という形になります。
尚、画像パッチは、deepな特徴における1pixelにて、よりロバストに代替されます。
複数フィルタによる多段な畳込み結果、及び、それらの共起という計算過程を経た、抽象的でリッチな代替です。
また、この代替は、ImageNetを識別するために抽出された特徴であるものの、概ね全般の課題感に適用可能というのが論文の主張です。
また、逆に言えば、画像パッチにおける複数pixel×複数pixelの特徴が、概ねdeepな特徴における1pixelに集約されます。
そして、その複数pixel×複数pixelの範囲が受容野となります。
(厳密には複雑な話になるので、それは一旦割愛させて頂きますが、PatchCoreの説明にてもう少しだけ触れさせて頂きます。)
もう少しだけ掘り下げさせて頂くと、そのような画像パッチの突合というアルゴリズム特性上、SPADEの異常位置セグメンテーションは、pixel依存がありません。
pixel依存とは、画像上のどこに何が写っているかという傾向までを学習してしまうことです。
つまり、少し画像を横や縦に平行移動しただけで、結果がブレてしまう傾向です。
SPADEの異常位置セグメンテーションは、その点、クエリ画像の全パッチと、K個のご近所さん画像の全パッチとの突合、即ち、距離計算を総当りで実施している為、対象が左上に写っていようが、右下に写っていようが、大まかにはロバストに対応できる想定となります。
この点は、SPADEが非常に使い勝手の良い点です。
ただし、先に課題感を指摘してしまいますが、K個のご近所さん画像の全パッチのみが、総当り距離計算対象というが、後続の論文PatchCoreにて指摘される点となります。
最終的な特徴ベクトルは、画像の全体傾向を高い抽象度で示すものですが、それを用いてピックアップをしたK個のご近所さん画像が、画像パッチ単位でも似た傾向のものを保持しているかというと、ちょっと疑問です。
例えば、黒猫が写っている画像の背景と、馬が写っている画像の背景が同様である場合には、突合の対象として、むしろ馬の画像をピックアップしたいところですが、背景の写り込みが少ない場合にはそうはいかず、背景の異なる黒猫がピックアップされることでしょう。
この突合対象の正常画像パッチに制限がかかってしまうことが、SPADEの異常位置セグメンテーションと課題感といいますか、限界となります。
ちなみに、実際にKの数を増やしていくと、SPADEの異常位置セグメンテーションの精度が上がっていく傾向があります。
これは、そういった背景からであることが推測されます。
また、いらずらにKの値を増やすしますと、突合対象が増えることから処理速後が長くなり、特徴ギャラリーが大きくなることからRAMが逼迫します。
逆に言えば、そういう背景があるからこそ、突合対象をK個のご近所さんに絞っているかと思われます。
尚、この課題感を突破すべくアプローチをしているのが、PatchCoreになるのですが、その説明は後程…。
さて、アルゴリズムの話に戻ります。
総当たりの距離計算が終わったら、その結果を集約します。
論文には、集約の仕方はKNNで、と書かれています。
具体的には、先程の結果から、 (特徴ギャラリーにある各pixel毎の特徴ベクトル本数:15,680, クエリ画像からの特徴テンソルの縦サイズ:56, クエリ画像からの特徴テンソルの横サイズ:56) という距離計算結果が得られていますが、これを1pixelずつ順に参照していきます。
1pixelずつには、15,680個の距離計算結果が存在しますが、これを小さい順に並べます。
そして、並べ替えた距離計算結果の小ささTOPKの平均を、その画像パッチの異常度合いとします。
これが、先述の となります。
尚、論文にて、MVTecでの実験においては、このKは1で実施したそうなので、15,680個の距離計算結果から最小のそれを1個得れば、K=1のKNNが実現できます。
それを実現するコードが以下となります。
# k nearest features from the gallery (k=1)
score_map = torch.min(dist_matrix, dim=0)[0]
print('score_map.shape =', score_map.shape)
plt.figure(figsize=(8, 6), dpi=100, facecolor='white')
plt.imshow(score_map, interpolation=None)
plt.colorbar()
plt.show()

これによって、 (クエリ画像からの特徴テンソルの縦サイズ:56, クエリ画像からの特徴テンソルの横サイズ:56) と同サイズの、画像パッチ別の異常スコアマップが作成されました。
図の色表現としては、K=1のKNNで得た特徴ギャラリーとの距離が、大きい箇所が黄色にて約3.5以上、小さい箇所が青色で約1.5以下という形になっています。
これが、元画像に対しての縮小版の異常スコアマップとなっています。
そして、この異常スコアマップを、元画像と同じ大きさにリサイズすることで、浅めの層の特徴 (Batch数:1, 特徴チャンネル数:256, 縦pixel数:56, 横pixel数:56) からの異常スコアマップ作成は完了となります。
或いは、浅めの層の特徴からの異常位置セグメンテーションが、完了となります。
特徴の拡大は、以下コードにて行います。
import torch.nn.functional as F
score_map = F.interpolate(score_map.unsqueeze(0).unsqueeze(0), size=224,
mode='bilinear', align_corners=False)
score_map = score_map[0, 0].cpu().numpy()
print('score_map.shape =', score_map.shape)
plt.figure(figsize=(8, 6), dpi=100, facecolor='white')
plt.imshow(score_map, interpolation=None)
plt.colorbar()
plt.show()

ぼんやり、「あの辺りに反応しているのかな…」と、当たりがつくかと思います。
これを画像と重ねて、確認をしてみましょう。
確認のコードが以下となります。
# https://github.com/gsurma/cnn_explainer/blob/main/utils.py#L16
def overlay_heatmap_on_image(img, heatmap, ratio_img=0.5):
img = img.astype(np.float32)
heatmap = 1 - np.clip(heatmap, 0, 1)
heatmap = cv2.applyColorMap((heatmap * 255).astype(np.uint8), cv2.COLORMAP_JET)
heatmap = heatmap.astype(np.float32)
overlay = (img * ratio_img) + (heatmap * (1 - ratio_img))
overlay = np.clip(overlay, 0, 255)
overlay = overlay.astype(np.uint8)
return overlay
img = [img_val[i] for i in np.where(y_val == 0)[0]][i_miss]
img_prep = img_prep_val[y_val == 0][i_miss]
plt.figure(figsize=(10, 3.6), dpi=100, facecolor='white')
plt.subplot(1, 2, 1)
plt.imshow(img_prep)
plt.subplot(1, 2, 2)
plt.imshow(score_map)
plt.colorbar()
plt.show()
plt.figure(figsize=(8, 8), dpi=100, facecolor='white')
score_map_ = score_map.copy()
score_map_ = score_map_ - np.min(score_map_)
score_map_ = score_map_ / np.max(score_map_)
plt.imshow(overlay_heatmap_on_image(img_prep, score_map_))
plt.show()


「なるほど、そこが異常か」という感じですね。
ちなみに、「学習データの猫画像の内、クエリ画像との距離の小さかった画像TOP5」を先程に確認しました。
それらの画像パッチの中に、似ている画像パッチがない部分が、赤く反応している形となります。
尚、繰り返しになりますが、これは「②画像上の異常位置セグメンテーション」での結果であり、「①画像単位の異常スコア算出」とは関係がありません。
特徴ギャラリー作成の際に、「①画像単位の異常スコア算出」での距離計算のためのKNN対象を用いていますが、関連はそれだけです。
つまり、「①画像単位の異常スコア算出」の要因分析となっていない点には注意が必要です。
一方で、「①画像単位の異常スコア算出」の処理の延長上に、「②画像上の異常位置セグメンテーション」の処理実現があります。
一連で異常位置まで検出できるのは、DN2からの発展と言えます。
さて、先程のこの異常スコアマップは、浅めの層からの実現である為、比較的局所的な特徴にて、パッチ合わせが行われています。
これが、中間の深さの層や、深めの層からとなると、それよりも大局的な特徴にて、パッチ合わせが行われる形になります。
最終的には、浅めの層、中間の深さの層、深めの層でそれぞれ算出した異常スコアマップを平均したものでもって、異常位置セグメンテーションが完了となります。
その実践に向けて、先程計算を行った、浅めの層からの異常スコアマップは、list変数に保存をしておきます。
score_map_mean = [] score_map_mean.append(score_map)
続いて、中間の深さの層から、異常スコアマップの算出を行います。
# https://github.com/mucunwuxian/SPADE-pytorch/blob/master/src/main.py#L130
# construct a gallery of features at all pixel locations of the K nearest neighbors
topk_feat_map = torch.from_numpy(f2_neighbor)
test_feat_map = torch.from_numpy(f2_query)
# adjust dimensions to measure distance in the channel dimension for all combinations
feat_gallery = topk_feat_map.transpose(1, 2).transpose(2, 3) # (K, C, H, W) -> (K, H, W, C)
feat_gallery = feat_gallery.flatten(0, 2) # (K, H, W, C) -> (KHW, C)
feat_gallery = feat_gallery.unsqueeze(1).unsqueeze(1) # (KHW, C) -> (KHW, 1, 1, C)
test_feat_map = test_feat_map.transpose(1, 2).transpose(2, 3) # (K, C, H, W) -> (K, H, W, C)
print('topk_feat_map.shape =', topk_feat_map.shape)
print('test_feat_map.shape =', test_feat_map.shape)
print('feat_gallery.shape =', feat_gallery.shape)
# calculate distance matrix
dist_matrix_list = []
for d_idx in tqdm(range(feat_gallery.shape[0] // 100 + 1)):
dist_matrix = torch.pairwise_distance(feat_gallery[d_idx*100:d_idx*100+100],
test_feat_map)
dist_matrix_list.append(dist_matrix)
dist_matrix = torch.cat(dist_matrix_list, 0)
print('dist_matrix.shape =', dist_matrix.shape)
# k nearest features from the gallery (k=1)
score_map = torch.min(dist_matrix, dim=0)[0]
print('score_map.shape =', score_map.shape)
score_map = F.interpolate(score_map.unsqueeze(0).unsqueeze(0), size=224,
mode='bilinear', align_corners=False)
score_map = score_map[0, 0].cpu().numpy()
print('score_map.shape =', score_map.shape)
img = [img_val[i] for i in np.where(y_val == 0)[0]][i_miss]
img_prep = img_prep_val[y_val == 0][i_miss]
plt.figure(figsize=(10, 3.6), dpi=100, facecolor='white')
plt.subplot(1, 2, 1)
plt.imshow(img_prep)
plt.subplot(1, 2, 2)
plt.imshow(score_map)
plt.colorbar()
plt.show()
plt.figure(figsize=(8, 8), dpi=100, facecolor='white')
score_map_ = score_map.copy()
score_map_ = score_map_ - np.min(score_map_)
score_map_ = score_map_ / np.max(score_map_)
plt.imshow(overlay_heatmap_on_image(img_prep, score_map_))
plt.show()
score_map_mean.append(score_map)


続いて、深めの層から、異常スコアマップの算出を行います。
# https://github.com/mucunwuxian/SPADE-pytorch/blob/master/src/main.py#L130
# construct a gallery of features at all pixel locations of the K nearest neighbors
topk_feat_map = torch.from_numpy(f3_neighbor)
test_feat_map = torch.from_numpy(f3_query)
# adjust dimensions to measure distance in the channel dimension for all combinations
feat_gallery = topk_feat_map.transpose(1, 2).transpose(2, 3) # (K, C, H, W) -> (K, H, W, C)
feat_gallery = feat_gallery.flatten(0, 2) # (K, H, W, C) -> (KHW, C)
feat_gallery = feat_gallery.unsqueeze(1).unsqueeze(1) # (KHW, C) -> (KHW, 1, 1, C)
test_feat_map = test_feat_map.transpose(1, 2).transpose(2, 3) # (K, C, H, W) -> (K, H, W, C)
print('topk_feat_map.shape =', topk_feat_map.shape)
print('test_feat_map.shape =', test_feat_map.shape)
print('feat_gallery.shape =', feat_gallery.shape)
# calculate distance matrix
dist_matrix_list = []
for d_idx in tqdm(range(feat_gallery.shape[0] // 100 + 1)):
dist_matrix = torch.pairwise_distance(feat_gallery[d_idx*100:d_idx*100+100],
test_feat_map)
dist_matrix_list.append(dist_matrix)
dist_matrix = torch.cat(dist_matrix_list, 0)
print('dist_matrix.shape =', dist_matrix.shape)
# k nearest features from the gallery (k=1)
score_map = torch.min(dist_matrix, dim=0)[0]
print('score_map.shape =', score_map.shape)
score_map = F.interpolate(score_map.unsqueeze(0).unsqueeze(0), size=224,
mode='bilinear', align_corners=False)
score_map = score_map[0, 0].cpu().numpy()
print('score_map.shape =', score_map.shape)
img = [img_val[i] for i in np.where(y_val == 0)[0]][i_miss]
img_prep = img_prep_val[y_val == 0][i_miss]
plt.figure(figsize=(10, 3.6), dpi=100, facecolor='white')
plt.subplot(1, 2, 1)
plt.imshow(img_prep)
plt.subplot(1, 2, 2)
plt.imshow(score_map)
plt.colorbar()
plt.show()
plt.figure(figsize=(8, 8), dpi=100, facecolor='white')
score_map_ = score_map.copy()
score_map_ = score_map_ - np.min(score_map_)
score_map_ = score_map_ / np.max(score_map_)
plt.imshow(overlay_heatmap_on_image(img_prep, score_map_))
plt.show()
score_map_mean.append(score_map)


浅めの層、中間の深さの層、深めの層から異常スコアマップが獲得できたら、それを平均した上で、最後にヒューリスティックな結果整形として、ガウシアンフィルターでなまして仕上げます。
from scipy.ndimage import gaussian_filter # average distance between the features score_map = np.mean(np.array(score_map_mean), axis=0) # apply gaussian smoothing on the score map score_map_ = gaussian_filter(score_map, sigma=4) plt.figure(figsize=(10, 3.6), dpi=100, facecolor='white') plt.subplot(1, 2, 1) plt.imshow(score_map) plt.colorbar() plt.subplot(1, 2, 2) plt.imshow(score_map_) plt.colorbar() plt.show() score_min = np.min(score_map_) score_max = np.max(score_map_) score_map__ = (score_map_ - score_min) / (score_max - score_min + 1e-10) plt.figure(figsize=(8, 8), dpi=100, facecolor='white') plt.imshow(overlay_heatmap_on_image(img_prep, score_map__)) plt.show()


これにて、SPADEのアルゴリズム実践は完了となります。
比較的シンプルながら、上手く目的が果たせているかと思います。
参考までに、animals10でも試してみます。
尚、あくまで参考までということで、実施コードは割愛させて頂きます。
DN2で画像レベルでの異常値が高かった、以下の画像で試してみようと思います。

このクエリ画像の、K=5のご近所さん画像は以下となります。





そして、浅い層での異常スコアマップが以下となります。


中間の層での異常スコアマップが以下です。


深い層での異常スコアマップが以下です。


最後に、上記3つのスコアマップの平均にて、異常位置セグメンテーションを行った結果が以下となります。


比較的上手くセグメンテーションができているようにも見えますが、画像パッチの不足から、例えば、猫の顔部分等、異常ではない箇所にもヒートマップが当たってしまっています。
データのアライメントが統制されていない場合における、SPADEでの異常位置セグメンテーションの限界が伺い知れる結果となっているかと思います。
MVTecでも試してみます。
MVTecでの試しについては、弊社謹製のSPADE-fastというリポジトリを使わせて頂きます。
このリポジトリは、SPADEを高速に実施すべく、全てのKNN処理に、faissライブラリを適用させて頂いているものとなっています。
READMEに沿って、処理を実施していただければ、例えば、 bottle データにて、以下の結果が得られます。
(結果1)

(結果2)

(結果3)

(結果4)

非常に良い結果となっていることが確認できます。
ちなみに、MVTecはむしろpixel依存をした方が精度が上がるくらいに整然としたデータです。
その為、SPADEの異常位置セグメンテーションについては、限られたご近所さんとのマッチング度合いが高く、より細部に対して、異常を示すことが可能となっています。
以上で、SPADEアルゴリズムについての解説を終えます。
DN2同様、シンプルな割に、非常に優秀なアルゴリズムであることが伺えたかと思います。
特に、コンピュータリソース、即ち、計算時間やRAM容量が、仮に無限であった場合には、SPADEの異常位置セグメンテーションは、無限の精度が発揮されるであろうことが妄想できます。
しかし、実際にはそれらは有限であり、無限となる将来も近くはないでしょう。
その為、ある種の割り切りを見せたり、RAM消費を抑えつつも極力多くのデータを保持するような方法が、後続の論文であるPaDiMとPatchCoreとでの展開となっていきます。
或いは、精度向上に向けた良き塩梅の1工夫なども発明がされていきます。
また、実は、SPADEの論文は「②画像上の異常位置セグメンテーション」を中心に語られており、「①画像単位の異常スコア算出」については、精度に関する記載がありません。
尚、先程紹介した弊社謹製のリポジトリに、その精度を測定結果を載せておりますが、実はデータの種類によっては、悪い数値が出てしまっています。
以下がその数値になります。

一方で、SPADEの「②画像上の異常位置セグメンテーション」については、以下のような素晴らしい精度が出ています。

ここには、実は大きな示唆があり、それは、ImageNetモデルの最終層の特徴ベクトルを使わない方が良いかもしれない、ということです。
DN2の論文では、どんなデータ種でも、DN2アプローチが概ね問題ないという主張でしたが、これが半分否定された形なのかと思います。
上手くいくものもあれば、上手くいかないものもあるという展開です。
一方で、SPADEの異常位置セグメンテーションは、全般上手く行っています。
また、SPADEの論文終盤にて、「selfsupervised feature learningを試してみたけど、ImageNetモデルを特徴抽出器に用いる方が精度が良かった」というような記載もあります。
このことから、ImageNetモデルが全面的に悪い訳ではなく、ImageNetモデルの最終層の特徴ベクトルを用いることが異常検知に向かなそう、ということが、MVTecを通した分析から見えてきます。
実際に、後続の論文、PaDiMとPatchCoreでは、最終層の特徴ベクトルが使われなくなるのですが、それはそれらの詳細説明にて紹介させて頂こうと思います。
おわりに
以上、今回の記事では、異常検知4手法の2つ目であるSPADEについて、詳細にまで踏み込んだ解説をさせて頂きました。
ここまで読んでくださった方には、またまた感謝です。🙇
次回の記事では、異常検知4手法の3つ目、PaDiMについて説明をさせて頂こうと思います。
そちらもお読み頂けますと、有り難い次第です。🙇
AnyTechでは、流体のためのAI「DeepLiquid」の研究/開発/事業に携わってくれる方を募集しております。
ご興味のある方はこちらから、是非お問い合わせください。